热门标签
热门文章
- 1智慧树考试怎么搜题找答案?用这八款神器就够了!!! #学习方法#微信_智慧树刷题
- 2python比较炫酷的代码,python漂亮gui界面模板
- 3使用深度学习进行语音情感识别:案例演示与代码实现_语音情感识别技术案例
- 4人工智能要在赋能实体经济上出实招
- 5astype()用法
- 6iOS开发-常用第三方开源框架介绍(你了解的ios只是冰山一角)_dszys.pro
- 7【论文笔记】CrossKD: Cross-Head Knowledge Distillation for Object Detection
- 8ARMV8-aarch64的虚拟内存(mmutlbcache)介绍-概念扫盲_armv8 arm64
- 9【C++庖丁解牛】哈希表/散列表的设计原理 | 哈希函数_c++中哈希表的原理
- 10家居建材商城|商家店铺|基于Springboot+Vue实现家居建材商城_vue商城商家端
当前位置: article > 正文
GWAS数据下载详解(2)_finngen数据库
作者:笔触狂放9 | 2024-07-08 00:17:40
赞
踩
finngen数据库
1、FinnGen数据库:Risteys FinnGen R11 + FinRegistry![]() https://risteys.finregistry.fi/
https://risteys.finregistry.fi/
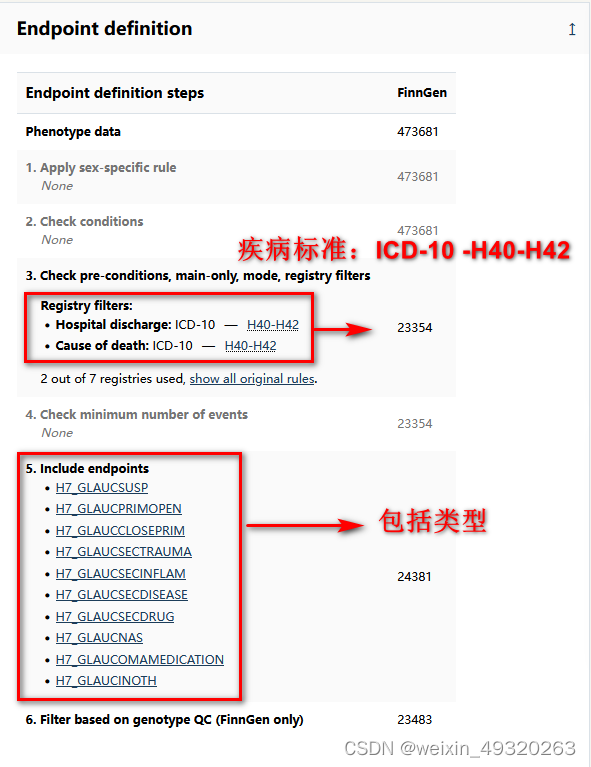
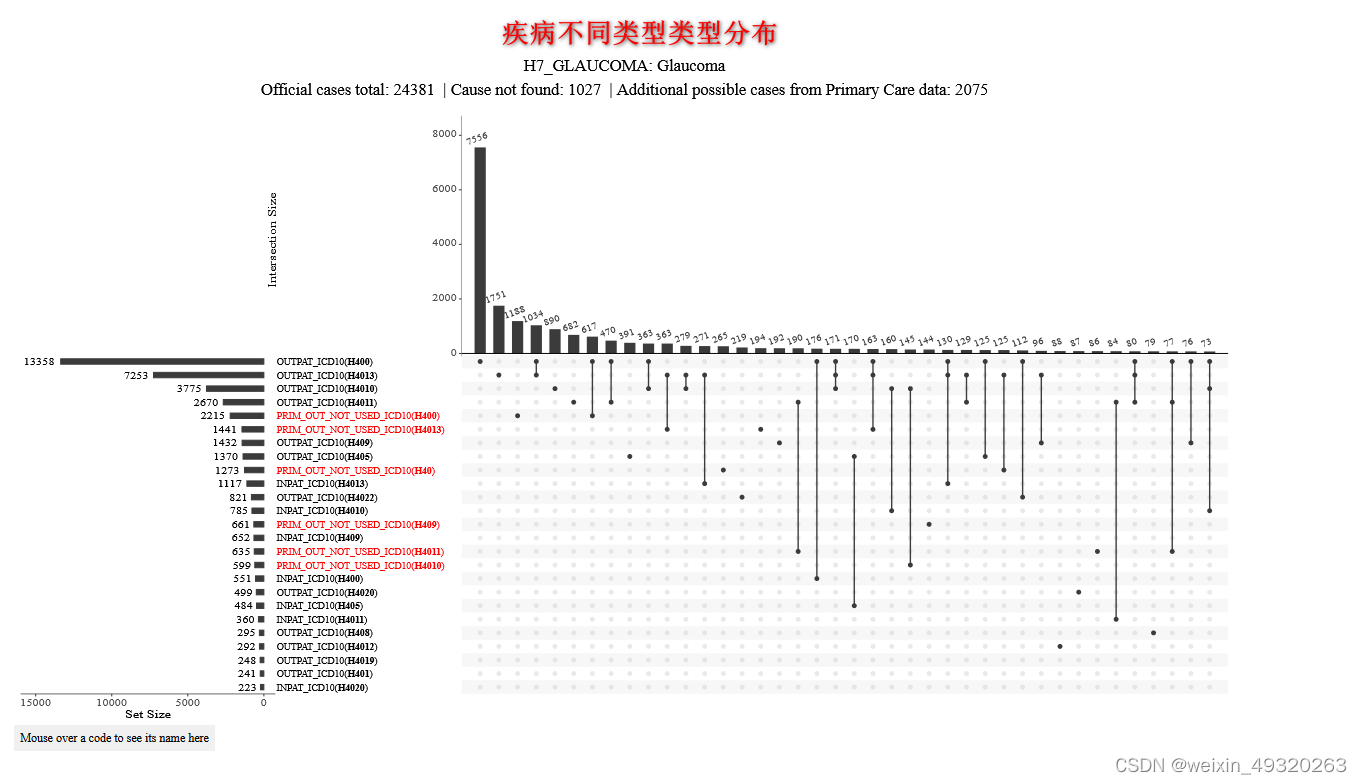
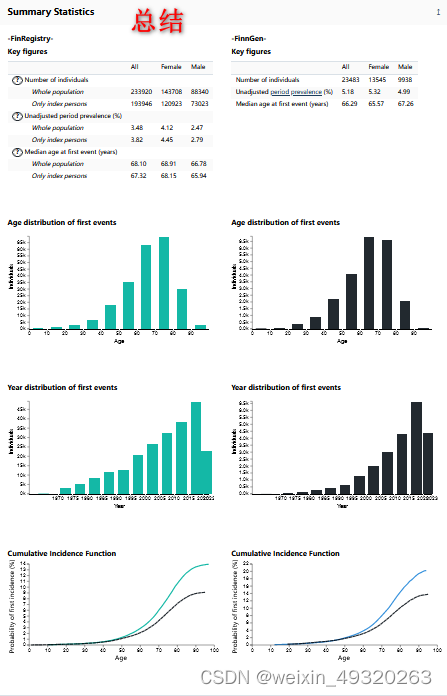
以检索"glaucoma"(青光眼)为例:https://risteys.finregistry.fi/


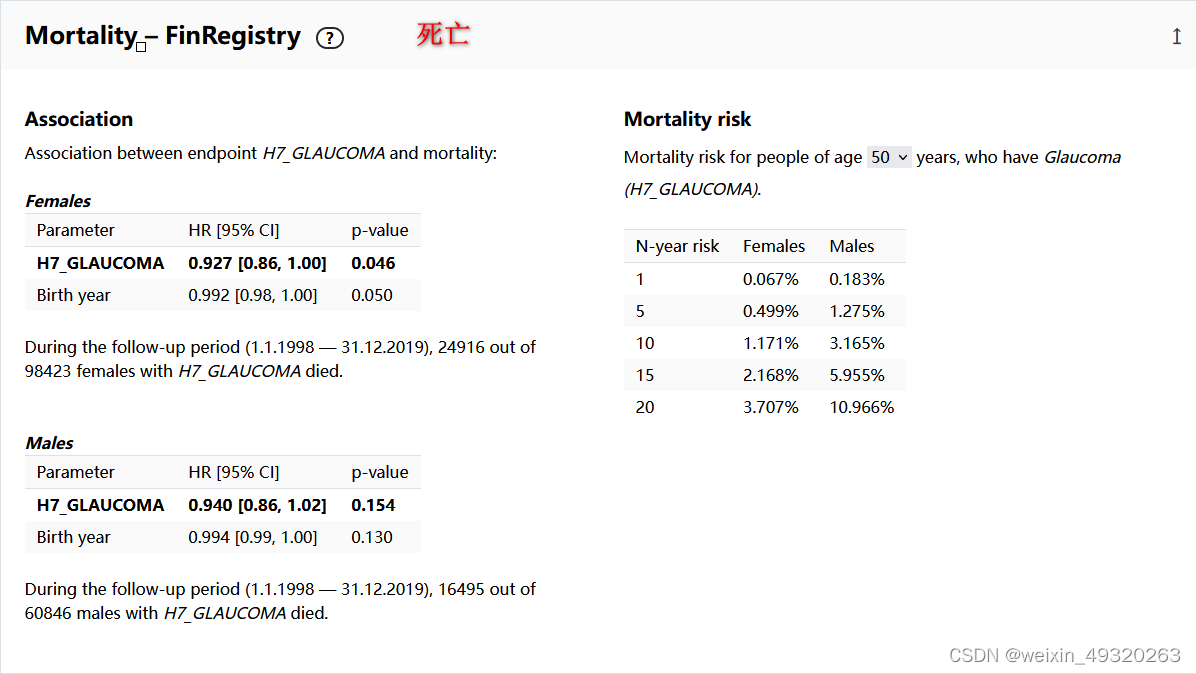

下载数据:
链接:https://storage.googleapis.com/finngen-public-data-r9/summary_stats/finngen_R11_H7_GLAUCOMA.gz
特点:R11代表数据库,H7_GLAUCOMA是endpoint name,其他数据可以替换后下载。
使用R语言整理数据:
"#chrom" :染色体;"pos"位置;"ref"对照等位基因;"alt":效应等位基因;"rsids":变量标识符;"nearest_genes":最近基因; "pval" p值;"mlogp" -log10P;"beta":效应大小;sebeta效应大小标准差;"af_alt":效应等位基因频率;"af_alt_cases":病例中的效应等位基因频率;"af_alt_controls":对照组中效应等位基因频率。
用TwoSampleMR整理出暴露数据和结局数据。
- #读取下载内容
- setwd("D:\\")#查看R语言当前工作路径,将txt文件放置给文件夹
- library('data.table')
- a <- fread("finngen_R9_O15_PRE_OR_ECLAMPSIA.gz",header = T)
- save(a,file="Finngen.RData")
- #获取数据变量
- colnames(a)
- #筛选强相关的变量:若5E-8筛选出来的变量较少,可适当调大P值(须有文献根据)
- ab<-subset(a,pval<5e-8)
- ab$phenotype<-"PRE_OR_ECLAMPSIA"
- #load("整理.RData")
- save(ab,file="整理.RData")
- #整理为TwoSampleMR所需要的双样本数据
- library(TwoSampleMR)
- #暴露数据
- exposure<-format_data(ab,
- type = "exposure",
- snp_col = "rsids",
- phenotype_col = "phenotype",
- beta_col = "beta",
- se_col = "sebeta",
- eaf_col="af_alt",
- effect_allele_col = "alt",
- other_allele_col = "ref",
- pval_col = "pval")
- #去除连锁不平衡(linkage disequilibrium)
- exposure_data<-clump_data(exposure,clump_r2 = 0.001)
-
- #结局数据
- outcome<-format_data(ab,
- snps=exposure_data$SNP,
- type = "outcome",
- snp_col = "rsids",
- phenotype_col = "phenotype",
- beta_col = "beta",
- se_col = "sebeta",
- eaf_col="af_alt",
- effect_allele_col = "alt",
- other_allele_col = "ref",
- pval_col = "pval")
整理出数据后即可进行分析。
声明:本文内容由网友自发贡献,不代表【wpsshop博客】立场,版权归原作者所有,本站不承担相应法律责任。如您发现有侵权的内容,请联系我们。转载请注明出处:https://www.wpsshop.cn/w/笔触狂放9/article/detail/797158
推荐阅读
相关标签


