热门标签
热门文章
- 1el-tree树形控件
- 2SmartUpload上传遇到Files' name is invalid or does not exist (1205).这是什么的错误_smartupload files name is invalid
- 3智慧工地管理平台APP源码基于物联网、云计算、大数据等技术
- 4C#对不同版本IIS的控制方法
- 5libcap库编译_如何编译libkcapi_1.1.5
- 6sqlserver中怎么查询字段为空的记录
- 7计算机毕业设计分享【使用java语言springboot框架/前台HTML+CSS 技术/MySQL】,springboot健康饮食推荐系统-49517(上万套实战教程,赠送源码)
- 8Springboot健康饮食推荐系统的设计与实现 49517 (免费领源码、附论文)可做计算机毕业设计JAVA、PHP、爬虫、APP、小程序、C#、C++、python、数据可视化、大数据、全套文案_基于spring boot的健康膳食推荐系统设计与实现参考文献
- 9MAC更改chromedriver版本_mac chrome如何切换版本
- 108.3-8.4搞机记录_华为多屏协同3168ac
当前位置: article > 正文
Docker 安装hadoop_docker 安装 hadoop
作者:你好赵伟 | 2024-03-08 17:17:41
赞
踩
docker 安装 hadoop
一、实验环境(实验设备)
硬件:单核CPU、内存1G 软件:Ubuntu 16.04操作系统、Docker、Hadoop
二、实验原理及内容
1.在Ubuntu系统中安装Docker
- Apt install docker
-
- 复制代码
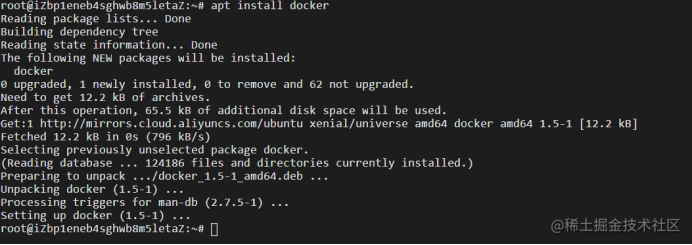
- Apt-get install docker.io
- 复制代码

2.Docker 安装 hadoop
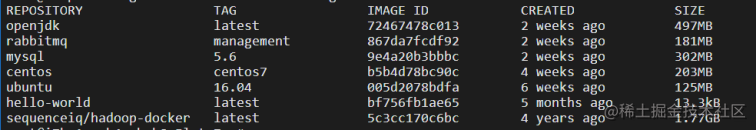
1).查找hadoop 镜像

2)将镜像pull下

3)通过docker images命令查看hadoop镜像是否pull成功
4)先通过命令
- docker run -i -t --name Master -h Master -p 50070:50070 sequenceiq/hadoop-docker /bin/bash
- 复制代码
运行一个hadoop镜像作为hadoop集群的namenode

再分别创建两个该hadoop集群的datanode
- docker run -i -t --name Slave1 -h Slave1 sequenceiq/hadoop-docker /bin/bash
- docker run -i -t --name Slave2 -h Slave2 sequenceiq/hadoop-docker /bin/bash
- 复制代码

这样hadoop的集群环境搭建完成。
3.hadoop三个节点之间无秘ssh配置
1)进入Master容器里面运行/etc/init.d/sshd start开启ssh,然后使用命令ssh-keygen -t rsa生成秘钥,最后将秘钥保存到authorized_keys中。

其余结点做相同操作。 进入3者查看ip信息 Master

Slave1

Slave2

4.hadoop的配置 由于hadoop集群环境已经搭建完成,我们只需更改Master节点上的hadoop配置文件,然后使用scp命令发送到其余各节点进行覆盖配置。 1)core-site.xml 配置

- <configuration>
- <property>
- <name>fs.defaultFS</name>
- <value>hdfs://had0:9000</value>
- </property>
- <property>
- <name>hadoop.tmp.dir</name>
- <value>/home/data/hadoopdata</value>
- </property>
- </configuration>
- 复制代码
2)hdfs-site.xml配置

- <property>
- <name>dfs.namenode.name.dir</name>
- <value>/home/data/hadoopdata/name</value>
- </property>
-   <!--配置存储namenode数据的目录-->
-   <property>
- <name>dfs.datanode.data.dir</name>
- <value>/home/data/hadoopdata/data</value>
- </property>
-   <!--配置存储datanode数据的目录-->
-   <property>
- <name>dfs.replication</name>
- <value>2</value>
- </property>
-   <!--配置部分数量-->
- 复制代码

3)mapred-site.xml配置

- <configuration>
- <property>
- <name>mapreduce.framework.name</name>
- <value>yarn</value>
- </property>  <!--配置mapreduce任务所在的资源调度平台-->
- </configuration>
- 复制代码
4)yarn-site.xml配置

- <configuration>
- <!-- Site specific YARN configuration properties -->
- <property>
- <name>yarn.resourcemanager.hostname</name>
- <value>slave1</value>
- </property>
- <!--配置yarn主节点-->
- <property>
- <name>yarn.nodemanager.aux-services</name>
- <value>mapreduce_shuffle</value>
- </property>
- <!--配置执行的计算框架-->
- </configuration>
- 复制代码
然后使用命令scp将该配置分发到其余两个节点,至此hadoop的配置完成

5.hadoop的运行 在Master中进入/usr/local/hadoop-2.7.0/sbin文件夹下执行命令./start-all.sh来启动该hadoop集群

成功启动之后使用命令jps查看是否启动

声明:本文内容由网友自发贡献,不代表【wpsshop博客】立场,版权归原作者所有,本站不承担相应法律责任。如您发现有侵权的内容,请联系我们。转载请注明出处:https://www.wpsshop.cn/w/你好赵伟/article/detail/211345
推荐阅读
相关标签


