- 1北航数据结构与程序设计查找与排序编程题
- 2Redis内存碎片问题_redis 碎片高风险
- 3测试人经验谈:需求不明确也能写出测试用例_测试用例没有经过评审,能给组员去使用吗
- 4秘密-安恒明御防火墙-入门信息安全教学
- 5基于51单片机的温度报警器设计
- 6github下载子文件夹_github网站dist目录怎么下载
- 7DevOps的原理及应用详解(五)
- 8大模型部署之前端页面编写框架,我选streamlit_大模型 流式输出 web页面
- 9【ArcGIS微课1000例】0120:ArcGIS批量修改符号的样式(轮廓)_批量制作arcgis符号库
- 10【Java】Eclipse的安装和JDK的安装与配置教程_eclipse配置jdk
【数学建模】聚类分析——python实现_python聚类分析
赞
踩
目录
一、储备知识
(1)何为聚类分析:
聚类分析又称为群分析,是研究问题的一种多元统计方法。聚类,就是聚集具有相似元素的集合成之为一类。一般来说存在着定性研究和定量研究以及相融汇的三种分析方法。通过选取共同指标,分析元素指标值之间的差距从而达到分类的目的。
(2)分类方法:
一般来说比较常用的是Q型分类(样品分类)和R型分类(指标分类),通俗来说前者是对样品进行聚类,后者是对变量进行聚类。此处重点讲解Q型分类,指标分类会在模糊聚类中说明
二、聚类分析的一般步骤
① 确定聚类类型:Q&R
② 数据预处理:使用标准化来克服不同的量纲的影响
标准化处理方法:(按矩阵列来处理)

③ 研究样品之间的相关关系:
1.两者度量方式(相似系数和距离)
相似系数:样品性质越接近,它们之间的相似系数的绝对值越接近与1;
计算方法:夹角余弦;相关系数等
距离:将每个样品看作p维空间上的一个点,通过计算任意两个样品之间的距离,可达到聚类的目的。因为距离越接近的点现归为一类,距离远的点暂时归为不同类。
常用距离:明科夫斯基距离;欧式距离;绝对值距离;切比雪夫距离;兰氏距离和马氏距离。那比较常用的是以下这些距离:

④ 计算距离矩阵或相似性系数矩阵D
由距离矩阵D,找到当前最小的D_i_j,并将类Gi合为一类得到一个新的类Gr={Gi、Gj},因为不太可能一次就聚类成功,因此要重新计算类间的距离,得到新的矩阵D,重复到全部合为一类。
三、聚类实操——python实现
事实上这里用R语言为更加简单。
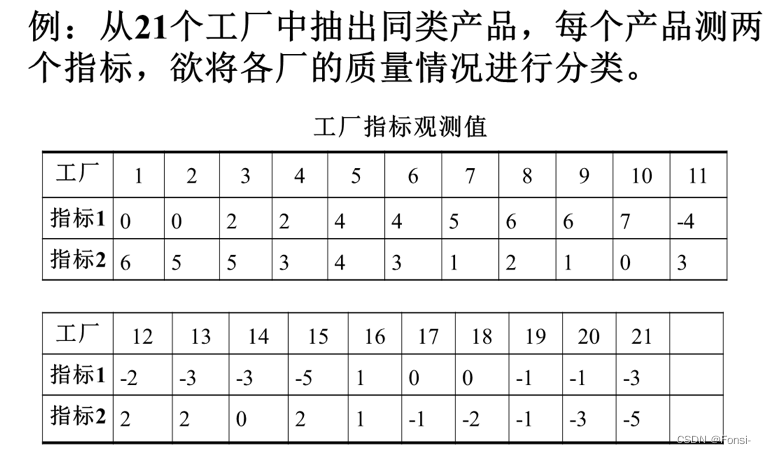
根据步骤,第一步本应该对其进行标准化处理,但事实上此处并不需要,因为各个指标本就在量纲上保持了统一了。但是根据流程也有必要对其进行标准化。
① 导入相关库
- import numpy as np
- from matplotlib import pyplot as plt
- from scipy.cluster.hierarchy import dendrogram,linkage
- import xlrd as xr
- import pandas as pd
- from sklearn import preprocessing
- from sklearn.cluster import AgglomerativeClustering
② 读取excel文件数据
- file_location="/Users/lifangjian/Desktop/聚类数据.xls"
- data=xr.open_workbook(file_location)
- sheet = data.sheet_by_index(0)
- lie=sheet.ncols
- hang=sheet.nrows
③ 形成数据矩阵,这里有很多处理方式,一种是常规的遍历法,另一种是列表推导式。默认第一行是变量名,比如指标A,指标B,第一列是样品名,比如工厂1,工厂2。
- #如果第一行是变量名,第一列是不同样本_遍历法
- datam=[]
- for i in range(1,hang):
- hanglie=[]
- for j in range(1,lie):
- hanglie.append(sheet.cell_value(i,j))
- datam.append(hanglie)
- print(datam)
- #列表推导式法_得到所有的值,按照行列排列,第一行j个数,第2行j个数。
- hanglie=[sheet.cell_value(i,j) for i in range(1,hang) for j in range(1,lie)] #得到所有ij的值
- stats = [[sheet.cell_value(r,c) for c in range(1,sheet.ncols)] for r in range(1,sheet.nrows)]#得到所有行列值
- print(stats)
- stats = pd.DataFrame(stats)
- print(stats)
-
- #如果第一行不同的变量,第一行是不同样本_遍历法,如果不是,建议在excel文件中使用=transpose命令
- lie=sheet.nrows
- hang=sheet.ncols
- sheet= data.sheet_by_index(1)
- datam=[]
- for j in range(1,lie):
- liehang=[]
- for i in range(1,hang):
- liehang.append(sheet.cell_value(i,j))
- datam.append(liehang)
- print(datam)
输出数据矩阵结果
[[0.0, 6.0], [0.0, 5.0], [2.0, 5.0], [2.0, 3.0], [4.0, 4.0], [4.0, 3.0], [5.0, 1.0], [6.0, 2.0], [6.0, 1.0], [7.0, 0.0], [-4.0, 3.0], [-2.0, 2.0], [-3.0, 2.0], [-3.0, 0.0], [-5.0, 2.0], [1.0, 1.0], [0.0, -1.0], [0.0, -2.0], [-1.0, -1.0], [-1.0, -3.0], [-3.0, -5.0]]
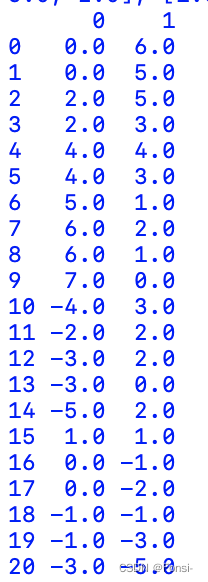
- stats_frame=pd.DataFrame(stats)
- normalizer=preprocessing.scale(stats_frame)
- stats_frame_nomalized=pd.DataFrame(normalizer)
- print(stats_frame)
- print(stats_frame_nomalized)
如果需要标准化,就按照以上的代码实现就可以,上面是Z分数标准化法。
④ 输出聚类过程
- z=linkage(stats,"average",metric='euclidean',optimal_ordering=True)
- print(z)
- # average=类平均法,ward=离差平方和法,sin=最短距离法,com=最长距离法,med=中间距离法,cen=重心法,fle=可变类平均法

第一列表示聚类类别,第二列表示和哪一类聚类,第三列是类之间的距离,第四列是簇中含有的类别的个数
这里对于各种方式的选取建议在IDLE中输入:
help(linkage)⑤ 画出动态聚类图
- fig, ax = plt.subplots(figsize=(10,9))
- dendrogram(z, leaf_font_size=14) #画图
- plt.title("Hierachial Clustering Dendrogram")
- plt.xlabel("Cluster label")
- plt.ylabel("Distance")
- plt.axhline(y=4) #画一条分类线
- plt.show()
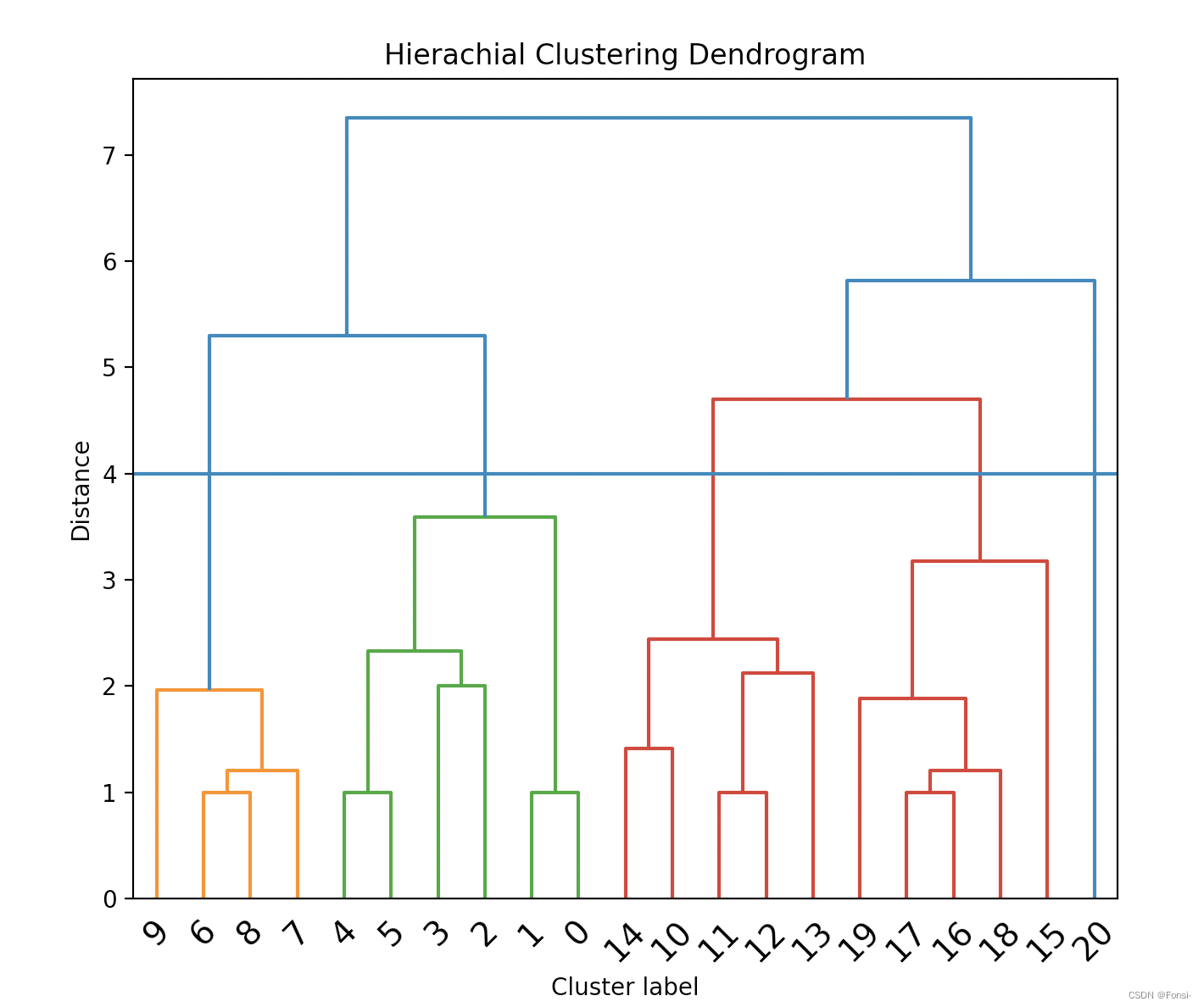
要注意,python里面第一个是0,因此此处正常阅读时要给Cluster label加1来看。
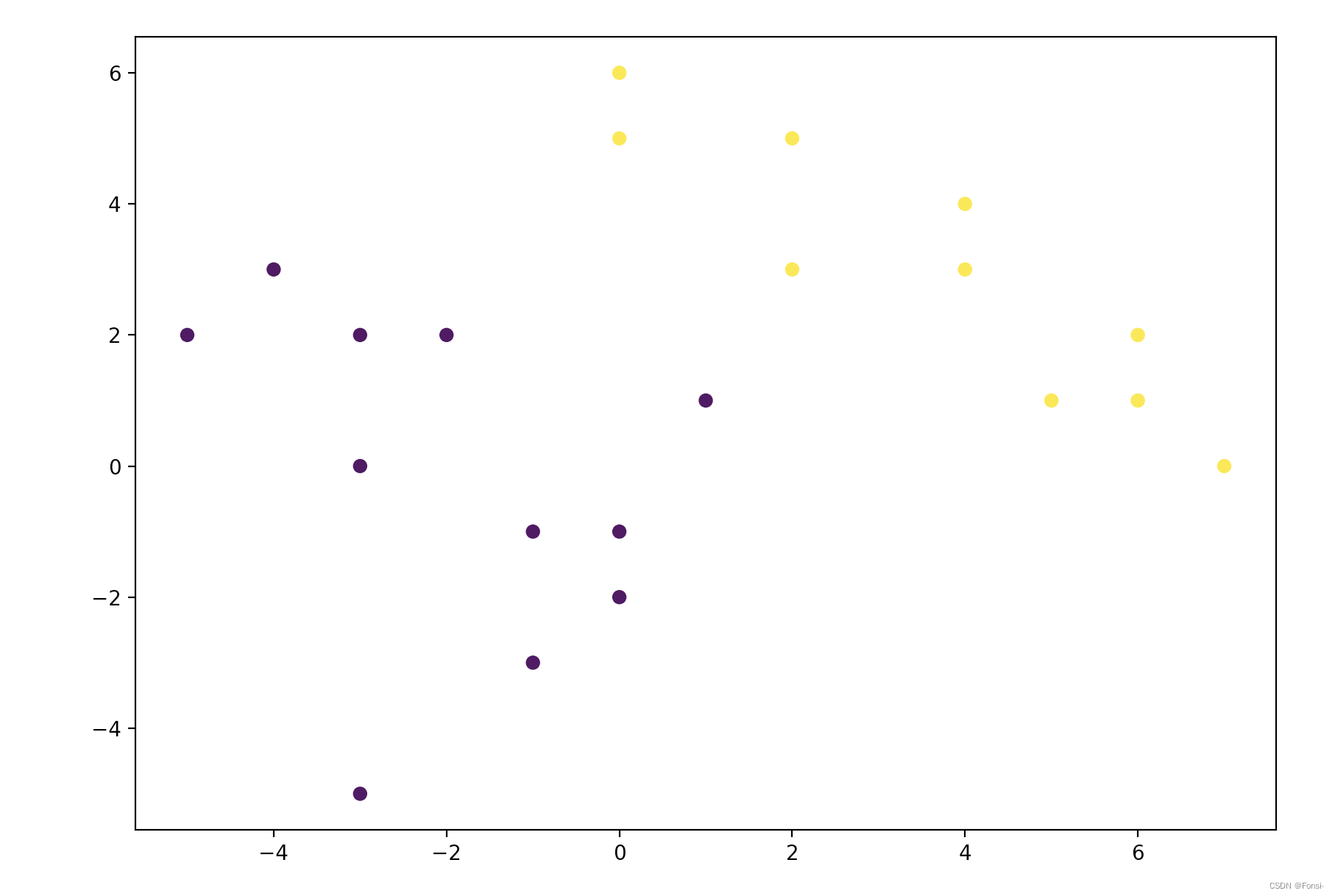
⑤ 画出聚类散点图,以2簇为例
- cluster = AgglomerativeClustering(n_clusters=2, affinity='euclidean', linkage='average')
- #linkage模式可以调整,n_cluser可以调整
- print(cluster.fit_predict(stats))
- plt.figure(figsize=(10, 7))
- plt.scatter(stats_frame[0],stats_frame[1], c=cluster.labels_)
- plt.show()
四、代码总结
- import numpy as np
- from matplotlib import pyplot as plt
- from scipy.cluster.hierarchy import dendrogram,linkage
- import xlrd as xr
- import pandas as pd
- from sklearn import preprocessing
- from sklearn.cluster import AgglomerativeClustering
- file_location="/Users/lifangjian/Desktop/聚类数据.xls"
- data=xr.open_workbook(file_location)
- sheet = data.sheet_by_index(0)
- lie=sheet.ncols
- hang=sheet.nrows
- #如果第一行是变量名,第一列是不同样本_遍历法
- datam=[]
- for i in range(1,hang):
- hanglie=[]
- for j in range(1,lie):
- hanglie.append(sheet.cell_value(i,j))
- datam.append(hanglie)
- print(datam)
- #列表推导式法_得到所有的值,按照行列排列,第一行j个数,第2行j个数。
- hanglie=[sheet.cell_value(i,j) for i in range(1,hang) for j in range(1,lie)] #得到所有ij的值
- stats = [[sheet.cell_value(r,c) for c in range(1,sheet.ncols)] for r in range(1,sheet.nrows)]#得到所有行列值
- stats_frame=pd.DataFrame(stats)
- normalizer=preprocessing.scale(stats_frame)
- stats_frame_nomalized=pd.DataFrame(normalizer)
- print(stats_frame)
- print(stats_frame_nomalized)
-
-
- z=linkage(stats,"average",metric='euclidean',optimal_ordering=True)
- print(z)
- # average=类平均法,ward=离差平方和法,sin=最短距离法,com=最长距离法,med=中间距离法,cen=重心法,fle=可变类平均法
- fig, ax = plt.subplots(figsize=(10,9))
- dendrogram(z, leaf_font_size=14) #画图
- plt.title("Hierachial Clustering Dendrogram")
- plt.xlabel("Cluster label")
- plt.ylabel("Distance")
- plt.axhline(y=4) #画一条分类线
- plt.show()
- cluster = AgglomerativeClustering(n_clusters=2, affinity='euclidean', linkage='average')
- #linkage模式可以调整,n_cluser可以调整
- print(cluster.fit_predict(stats))
- plt.figure(figsize=(10, 7))
- plt.scatter(stats_frame[0],stats_frame[1], c=cluster.labels_)
- plt.show()


