热门标签
热门文章
- 1WakeLock小计_acquire_causes_wakeup
- 2[题解]《算法零基础100讲》(第25讲) 字符串算法(五) - 字符串反转_char *reverseprefix(char *word, char ch)在主函数里怎么表示调
- 3linux操作系统进程阻塞原理_操作系统如何做到进程阻塞的
- 4MySQL+MySQLWorkBench安装和配置_下载mysqlworkbench并配置
- 5C#微信公众号自动回复源码_c# 微信实现自动回复
- 6python_要求实现fun1函数,身高和体重作为参数,输出肥胖状况。 编写代码实现。结果保留两
- 7CCF编程能力等级认证GESP—C++4级—20230923_下列关于c++语 中指针的叙述 ,不正确的是( )。 可以定义指向int类型的指针。
- 8【笔记】动⼿学深度学习(花书)|| Aston Zhang Mu Li Zachary C. LiptonAlexander J. Smola
- 9【岗位详情】腾讯广告后台开发工程师(北京)_来这里你将收获广阔的平台
- 10中兴ZXVb860av2.1t刷机固件,芯片晶晨S905l-b,不失效线刷包,当贝桌面
当前位置: article > 正文
使用RDKit对分子进行聚类_rdkit结构相似性聚类
作者:小蓝xlanll | 2024-03-31 07:34:42
赞
踩
rdkit结构相似性聚类
- from rdkit import Chem
- from rdkit.Chem import AllChem
-
- def ClusterFps(fps,cutoff=0.2):
- from rdkit import DataStructs
- from rdkit.ML.Cluster import Butina
-
- # first generate the distance matrix:
- dists = []
- nfps = len(fps)
- for i in range(1,nfps):
- sims = DataStructs.BulkTanimotoSimilarity(fps[i],fps[:i])
- dists.extend([1-x for x in sims])
-
- # now cluster the data:
- cs = Butina.ClusterData(dists,nfps,cutoff,isDistData=True)
- return cs

RDKit实现了Butina聚类算法,基于文献 `Unsupervised Database Clustering Based on Daylight's Fingerprint and Tanimoto Similarity: A Fast and Automated Way to Cluster Small and Large Data Sets', JCICS, 39, 747-750 (1999)`
上面定义好了函数,接下来读取化合物。
- ms = [x for x in Chem.SDMolSupplier('ZINC_example.sdf')]
- print (len(ms))
- output: 10000
计算指纹并聚类,cutoff是指Tanimoto 相似度的阈值 ,差异度大于cutoff的化合物会被分到不同的类别中。
- fps = [AllChem.GetMorganFingerprintAsBitVect(x,2,1024) for x in ms
- clusters=ClusterFps(fps,cutoff=0.4)
- print(clusters[100])
- output: (553, 297, 547)
- from rdkit.Chem import Draw
- from rdkit.Chem.Draw import IPythonConsole
- m1 = ms[553]
- m2 = ms[297]
- m3 = ms[547]
- mols=(m1,m2,m3)
- Draw.MolsToGridImage(mols)
cluster[100] 中有三个分子,分别是553,297,547号。使用Draw函数绘制如下:
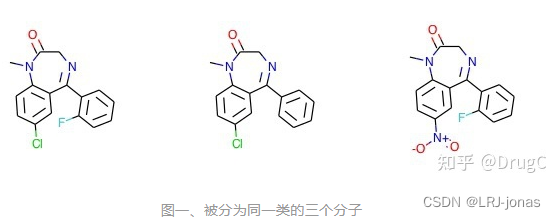
RDkit的聚类算法可以支持10万级别的化合物库,然而在内存为125 GB的服务器上测试20万分子的聚类时,可以看到随着聚类的时间增长,占用的内存也不停增加,最终在半个小时后,聚类20万分子由于内存溢出而崩溃。
声明:本文内容由网友自发贡献,不代表【wpsshop博客】立场,版权归原作者所有,本站不承担相应法律责任。如您发现有侵权的内容,请联系我们。转载请注明出处:https://www.wpsshop.cn/w/小蓝xlanll/article/detail/343697
推荐阅读
相关标签


