热门标签
热门文章
- 1ONLYOFFICE 桌面编辑器 8.1重磅来袭:全新功能提升您的办公效率_onlyoffice8.1界面图片
- 2【DL】《【Chatgpt】+【simplelatex】再也不用愁数学公式!!!》
- 3Packet Tracer模拟一次简单的HTTP请求_packettracer 发送请求
- 4史上最全:python自动化测试栈,确定好你从功能测试后的进阶方向_软件测试之python全栈自动化测试工程师工具包
- 5yolov5驾驶员不规范行为检测_驾驶员行为检测 csdn
- 6最全Java+MySQL实现学生管理系统_用java和mysql做一个简单的管理系统,Spring都没弄明白凭什么拿高薪_java+mysql学生管理系统
- 7弹性公网IP带宽与内网带宽有何差异?_为什么出网带宽使用量大于弹性公网ip带宽
- 8数据中台架构体系浅析_工厂数据体系
- 9AI大模型研究框架|附28页PDF文件下载_ai大模型.pdf
- 10【微信小程序-原生开发】启动时自动升级更新到最新版本_微信小程序发布新版本后多久更新
当前位置: article > 正文
层次聚类分析
作者:weixin_40725706 | 2024-07-01 01:33:15
赞
踩
层次聚类分析
1、python语言
- from scipy.cluster import hierarchy # 导入层次聚类算法
- import matplotlib.pylab as plt
- import numpy as np
-
- # 生成示例数据
- np.random.seed(0)
- data = np.random.random((20,1))
-
- # 使用树状图找到最佳聚类数
- Z = hierarchy.linkage(data,method='weighted',metric='euclidean')
- re = hierarchy.dendrogram(Z,color_threshold=0.2,above_threshold_color='#bcbddc')
-
- # 输出节点标签
- print(re["ivl"])
-
- # 画图
- plt.title('Dendrogram') # 标题
- plt.xlabel('Customers') # 横标签
- plt.ylabel('Euclidean distances') # 纵标签
- plt.show()
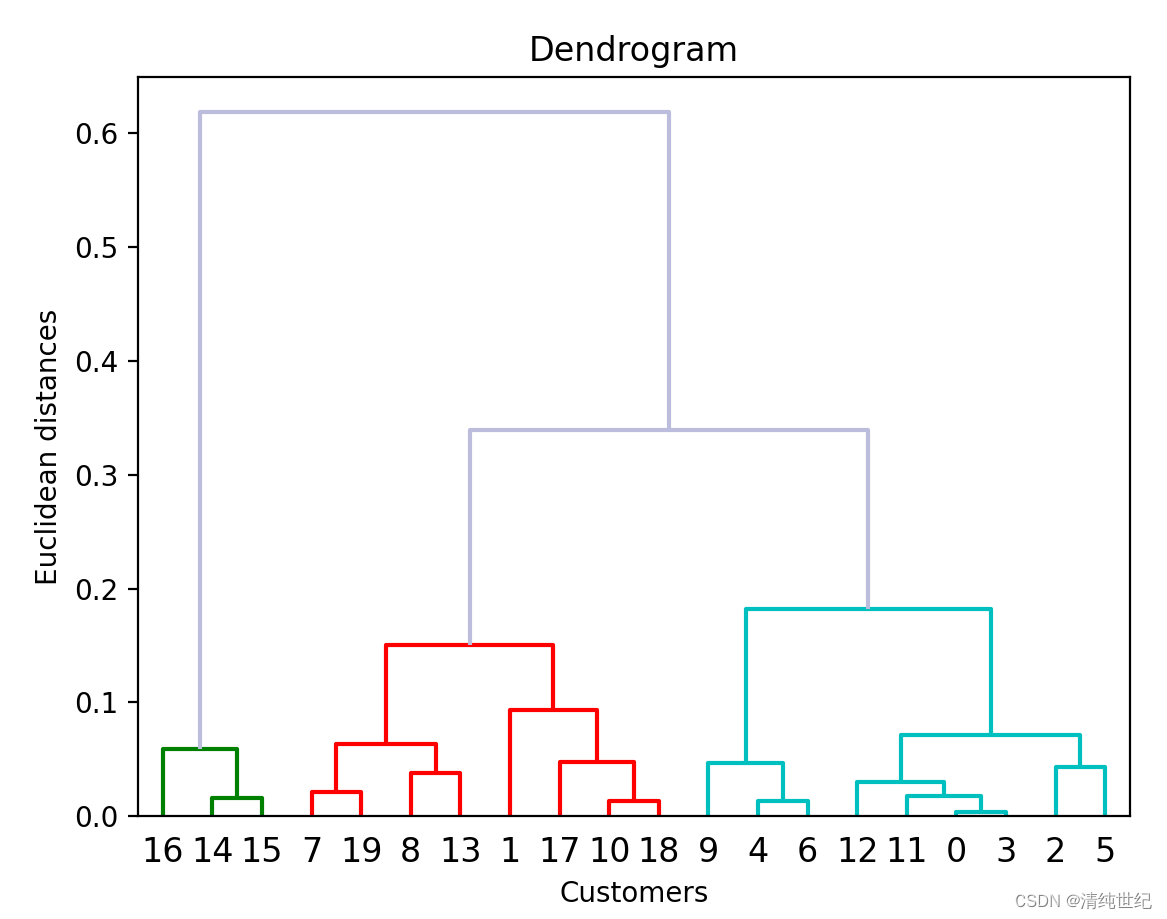
dendrogram函数参数:
- Z:层次聚类的结果,即通过scipy.cluster.hierarchy.linkage()函数计算得到的链接矩阵。
- p:要显示的截取高度(y轴的阈值),可以用于确定划分群集的横线位置。
- truncate_mode:指定截取模式。默认为None,表示不截取,可以选择 'lastp' 或 'mlab' 来截取显示。
- labels:数据点的标签,以列表形式提供。
- leaf_font_size:叶节点的字体大小。
- leaf_rotation:叶节点的旋转角度。
- show_leaf_counts:是否显示叶节点的数量。
- show_contracted:是否显示合并的群集。
- color_threshold:显示不同颜色的阈值,用于将不同群集算法聚类为不同颜色。
- above_threshold_color:超过阈值的线段颜色。
- orientation:图形的方向,可以选择 'top'、'bottom'、'left' 或 'right'。
hierarchy.linkage参数:
- y:输入的数据集,可以是一个离散的样本点的集合,或者是一个已经计算好的距离矩阵。
-
- method:指定层次聚类的算法,常用的方法包括 “single”(最近邻),“complete”(最远邻),“average”(平均距离),“weighted”(加权平均法),默认为 “single”。
-
- metric:指定用于计算距离的方法,常见的包括 “euclidean”(欧氏距离),“manhattan”(曼哈顿距离),“cosine”(余弦相似度)等,默认为 “euclidean”。
-
- optimal_ordering:是否对连接矩阵进行优化,以获得更好的划分,默认为 False。
-
- pooling_func:当 y 是浮点型矩阵时,指定汇聚的方法,默认为 np.mean,即使用平均值。
假设我们输出Z值,获得以下结果:
- from scipy.cluster import hierarchy # 导入层次聚类算法
- import numpy as np
- import pandas as pd
-
- # 生成示例数据
- np.random.seed(0)
- data = np.random.random((8,1))
-
- # 使用树状图找到最佳聚类数
- Z = hierarchy.linkage(data,method='weighted',metric='euclidean')
- row_dist_linkage = pd.DataFrame(Z,
- columns=['Row Label 1','Row Label 2','Distance','Item Number in Cluster'],
- index=['Cluster %d' % (i+1) for i in range(Z.shape[0])])
- print("\nData Distance via Linkage: \n",row_dist_linkage)

其中,第一列和第二列代表节点标签,包含叶子和枝子;第三列代表叶叶(或叶枝,枝枝)之间的距离;第四列代表该层次类中含有的样本数(记录数)。注:因此,我们可以第三列距离结合图来确定不同簇的样本数量。这里的数量为(n-1),即样本总数减1。
另外一种方法(注意:sklearn必须是最新版,我的是0.24.2---之前我的是0.19的,否则报错):
sklearn更新升级:
pip install -U scikit-learn -i https://pypi.tuna.tsinghua.edu.cn/simple- import numpy as np
- from scipy.cluster.hierarchy import dendrogram
- from sklearn.cluster import AgglomerativeClustering
- import matplotlib.pyplot as plt
-
- X = np.random.random((10,1))
-
- def plot_dendrogram(model, **kwargs):
- # 创建链接矩阵,然后绘制树状图
- # 创建每个节点的样本计数
- counts = np.zeros(model.children_.shape[0])
- n_samples = len(model.labels_)
- for i, merge in enumerate(model.children_):
- current_count = 0
- for child_idx in merge:
- if child_idx < n_samples:
- current_count += 1 # 叶子节点
- else:
- current_count += counts[child_idx-n_samples]
- counts[i] = current_count
- linkage_matrix = np.column_stack([model.children_, model.distances_,
- counts]).astype(float)
- print(linkage_matrix)
- # 绘制相应的树状图
- dendrogram(linkage_matrix, **kwargs)
-
- # 设置 distance_threshold = 0 ,以确保我们计算的是完整的树
- model = AgglomerativeClustering(distance_threshold=0, n_clusters=None)
- model = model.fit(X)
-
- plt.title('Hierarchical Clustering Dendrogram')
- # 绘制树状图的前三个级别
- plot_dendrogram(model, truncate_mode='level', p=3)
- plt.xlabel("Number of points in node (or index of point if no parenthesis).")
- plt.show()
2、R语言
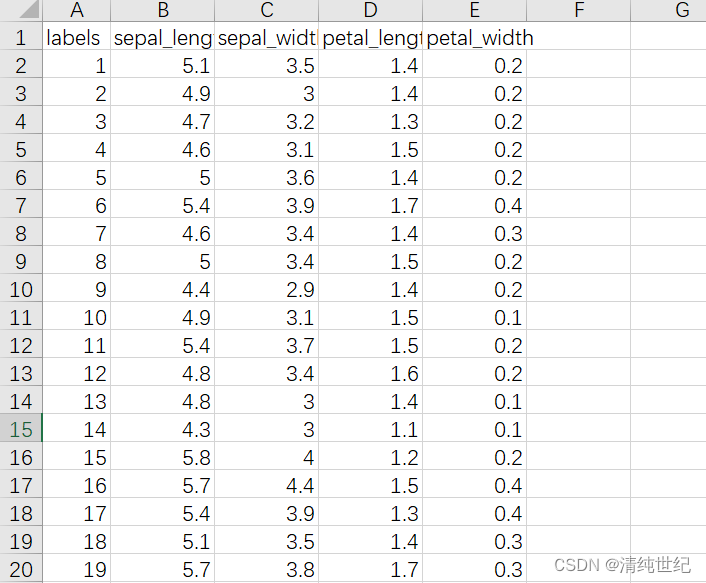
数据:iris.zip
- setwd("D:/Desktop/0000/R") #更改路径
-
- df <- read.csv("iris.csv",header = T, row.names = 1) #读取工作路径文件
- head(df) #查看前6行
- hc <- hclust(dist(df))
-
- library(ggtree)
-
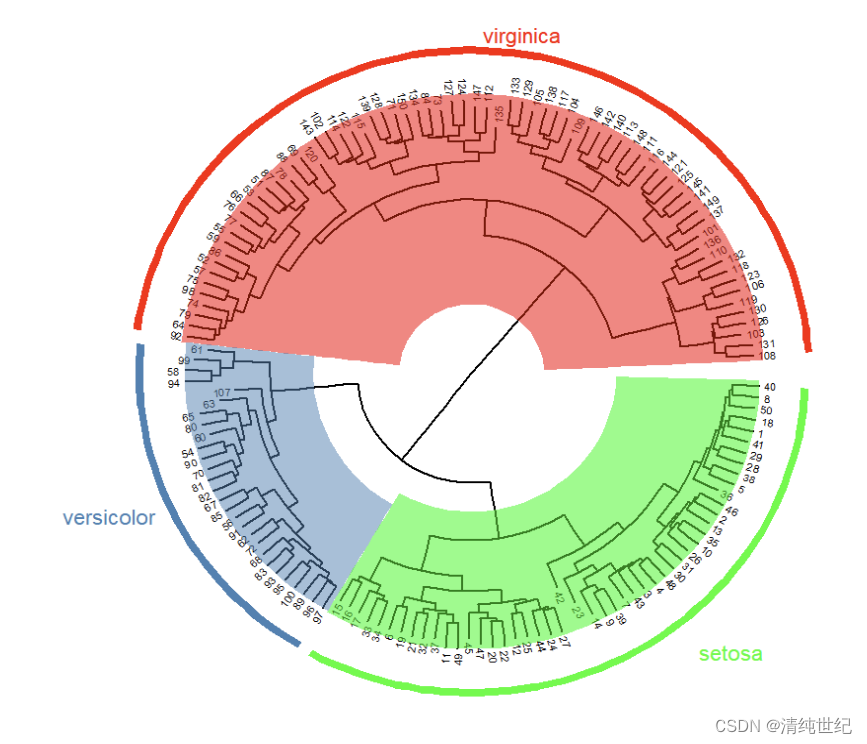
- ggtree(hc,layout="circular",branch.length = "daylight")+
- xlim(NA,3)+
- geom_tiplab2(offset=0.1,
- size=2)+
- #geom_text(aes(label=node))+
- geom_highlight(node = 152,fill="red")+
- geom_highlight(node=154,fill="steelblue")+
- geom_highlight(node=155,fill="green")+
- geom_cladelabel(node=152,label="virginica",
- offset=1.2,barsize = 2,
- vjust=-0.5,color="red")+
- geom_cladelabel(node=154,label="versicolor",
- offset=1.2,barsize = 2,
- hjust=1.2,color="steelblue")+
- geom_cladelabel(node=155,label="setosa",
- offset=1.2,barsize = 2,
- hjust=-1,color="green")
如果没有安装ggtree则先安装
- install.packages("BiocManager")
- BiocManager::install('ggtree')
-
- 当然,我们可以指定版本安装:BiocManager::install('ggtree',version = "3.17")
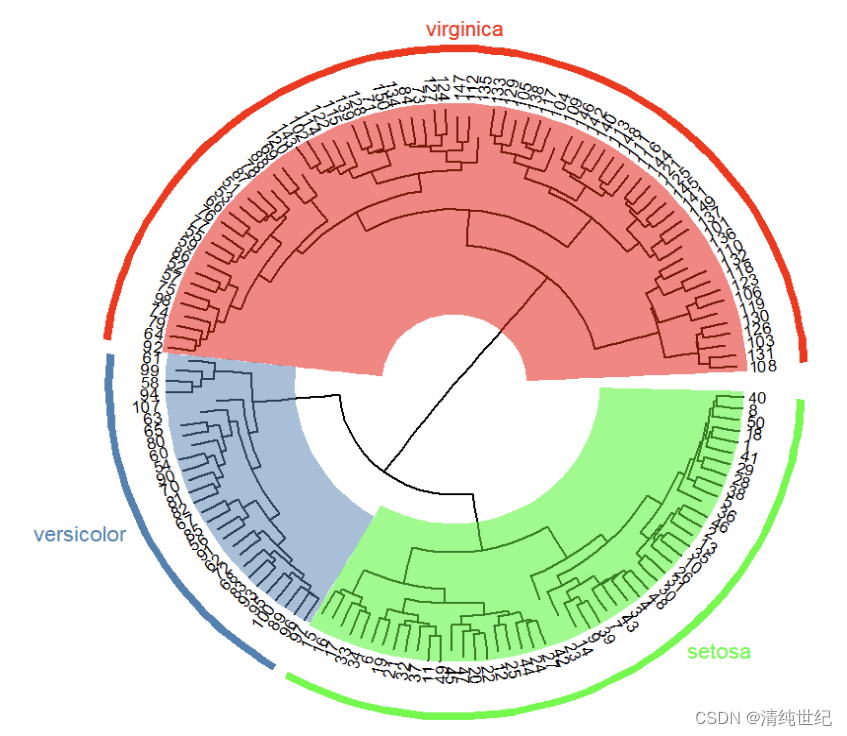
对上面代码在修改下:
- setwd("D:/Desktop/0000/R") #更改路径
-
- df <- read.csv("iris.csv",header = T, row.names = 1) #读取工作路径文件
- head(df) #查看前6行
- hc <- hclust(dist(df))
-
- library(ggtree)
- help(package="ggtree")
- ggtree(hc,layout="circular",branch.length=5,size = 0.5)+ #size = 0.5 线宽
- xlim(NA,3)+
- #theme_tree2()+ #显示x坐标范围
- geom_tiplab2(size=3,align=T,linesize = -0.0,linetype = 0,offset = 0.0001)+ #size = 3 标签大小
- #align=T 标签右对齐 linesize = 16 标签右对齐后会有线连接,
- #设置线的粗细 linetype = 1 设置线的类型,默认是虚线 offset=2设置标签距离枝末端的距离
- #geom_text(aes(label=node))+
- geom_highlight(node = 152,fill="red",
- #extendto = 0.05, #延长
- #extend =-0, #反向延长
- #alpha = 0.2
- )+
- geom_highlight(node=154,fill="steelblue")+
- geom_highlight(node=155,fill="green")+
- geom_cladelabel(node=152,label="virginica",
- offset=1.5,barsize = 2,
- vjust=-0.8,hjust=0.5,color="red")+
- geom_cladelabel(node=154,label="versicolor",
- offset=1.5,barsize = 2,
- hjust=1.2,color="steelblue")+
- geom_cladelabel(node=155,label="setosa",
- offset=1.5,barsize = 2,
- hjust=-1,vjust=-1,color="green")#+
- # #另外一种分类条带的方法
- # geom_strip(51,#起点,设置的是外节点
- # 114,#终点
- # label= "cluster 2", #分类标签名
- # offset= 1.5, #条带的偏移量
- # offset.text = 3, #标签的偏移量
- # barsize= 2, #条带宽度
- # #extend= 0.2, #延长条带的长度(两端)
- # color= "#9467BDFF", #条带颜色
- # angle= 90,#标签旋转角度
- # hjust= "center"
- # )
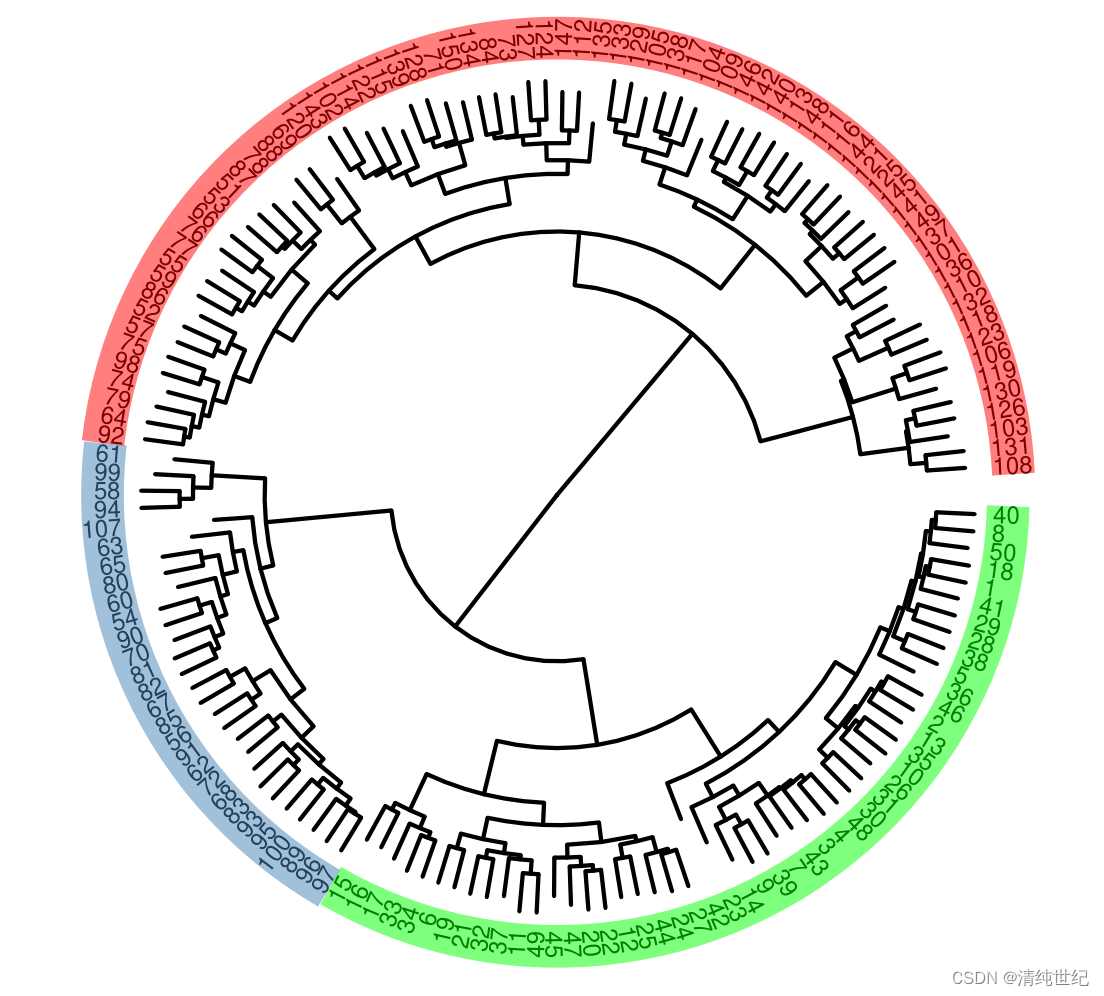
又或者:
- setwd("D:/Desktop/0000/R") #更改路径
-
- df <- read.csv("iris.csv",header = T, row.names = 1) #读取工作路径文件
- head(df) #查看前6行
- hc <- hclust(dist(df))
-
- library(ggtree)
- help(package="ggtree")
- ggtree(hc,layout="circular",branch.length=5,size = 0.5)+ #size = 0.5 线宽
- xlim(NA,3)+
- #theme_tree2()+ #显示x坐标范围
- geom_tiplab2(size=2,align=T,linesize = -0.0,linetype = 0,offset = 0.2)+ #size = 3 标签大小
- #align=T 标签右对齐 linesize = 16 标签右对齐后会有线连接,
- #设置线的粗细 linetype = 1 设置线的类型,默认是虚线 offset=2设置标签距离枝末端的距离
- #geom_text(aes(label=node))+
- geom_cladelab(node=152,label="",
- barcolor="red",
- barsize = 5,
- extend=0.5,
- offset=0.6,
- alpha = 0.5)+
- geom_cladelab(node=154,label="",
- barcolor="steelblue",
- barsize = 5,
- extend=0.5,
- offset=0.7,
- alpha = 0.5)+
- geom_cladelab(node=155,label="",
- barcolor="green",
- barsize = 5,
- extend=0.5,
- offset=0.6,
- alpha = 0.5)
除了上面这种方式外,我们还可以使用下面的方式获取(节点对齐):
- setwd("D:/Desktop/0000/R") #更改路径
- library(dendextend) #install.packages("dendextend")
- library(circlize) #install.packages("circlize")
-
- df <- read.csv("iris.csv",header = T, row.names = 1) #读取工作路径文件
- head(df) #查看前6行
- aa <- hclust(dist(df))
-
- # 设置画布大小为4英寸宽,4英寸高
- par(mar = c(4, 4, 2, 2) + 0.1)
- png("output.png", width = 4, height = 4, units = "in", res = 600)
-
- hc <- as.dendrogram(aa) %>%
- set("branches_lwd", c(1.5)) %>% # 线条粗细
- set("labels_cex", c(.9)) # 字体大小
-
- # 颜色
- hc <- hc %>%
- color_branches(k = 10) %>% #树状分支线条颜色
- color_labels(k = 10) #文字标签颜色
-
- # Fan tree plot with colored labels
- circlize_dendrogram(hc,
- labels_track_height = NA,
- dend_track_height = 0.7)
- # 结束绘图并关闭设备
- dev.off()
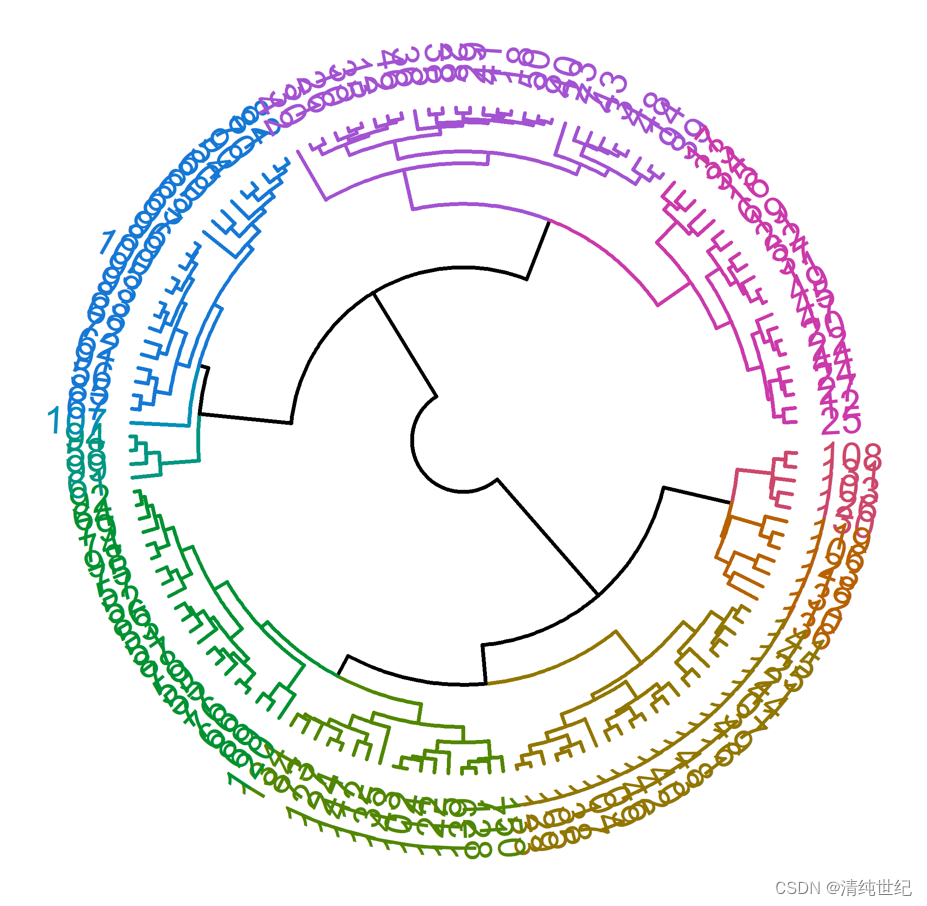
文件数据样式:
更多学习视频:【R包使用】ggtree美化树状图_哔哩哔哩_bilibili、树状图展示聚类分析的结果_哔哩哔哩_bilibili
3、密度聚类(DBSCAN)

简单例子:
下面是KMeans、层次聚类的结果:

那么使用密度聚类结果为:

代码:
- from sklearn.cluster import KMeans
- import matplotlib.pyplot as plt
- from sklearn.cluster import AgglomerativeClustering
- from sklearn.cluster import DBSCAN
- import numpy as np
-
- # 创建虚拟数据
- x1 = np.random.random((100,1))*10
- x2 = np.random.random((100,1))*1
- X = np.concatenate((x1,x2),axis=0)
-
- f,(ax1, ax2, ax3, ax4) = plt.subplots(1, 4, figsize=(8, 3))
-
- # KMeans
- km = KMeans(n_clusters=2).fit(X) # n_clusters=None, n_init=1, random_state=0
- y_km = km.labels_
- for i in range(max(y_km+1)):
- ax1.scatter(np.array([i for i in range(len(X))])[y_km==i],X[y_km==i], marker='o', s=40, label='Cluster '+str(i))
- ax1.set_title('K-means')
- ax1.set_xlabel('Feature 1')
- ax1.set_ylabel('Feature 2')
-
- # 层次聚类
- ac = AgglomerativeClustering().fit(X)
- y_ac = ac.labels_
- for i in range(max(y_ac+1)):
- ax2.scatter(np.array([i for i in range(len(X))])[y_ac==i],X[y_ac==i], marker='o', s=40, label='Cluster '+str(i))
- ax2.set_title('Agglomerative')
- ax2.set_xlabel('Feature 1')
- ax2.set_ylabel('Feature 2')
-
- # 密度聚类
- # eps 就是半径,min_samples就是 MinPts 值
- db = DBSCAN(min_samples=10).fit(X) # eps=0.2, min_samples=3, metric='euclidean'
- y_db = db.labels_
- for i in range(max(y_db+1)):
- ax3.scatter(np.array([i for i in range(len(X))])[y_db==i],X[y_db==i], marker='o', s=40, label='Cluster '+str(i))
- ax3.set_title('DBSCAN')
- ax3.set_xlabel('Feature 1')
- ax3.set_ylabel('Feature 2')
-
- # 原始数据
- ax4.scatter(np.array([i for i in range(len(X))])[:100], X[:100],c='lightblue',edgecolor='black', marker='o', s=40, label='Cluster 1')
- ax4.scatter(np.array([i for i in range(len(X))])[100:], X[100:], c='red',edgecolor='black', marker='s', s=40, label='Cluster 2')
- ax4.set_title('Original data')
- ax4.set_xlabel('Feature 1')
- ax4.set_ylabel('Feature 2')
-
- plt.legend()
- plt.tight_layout()
- plt.show()

我通过创建三个不同的聚类中心数据:
- from sklearn.cluster import KMeans
- import matplotlib.pyplot as plt
- from sklearn.cluster import AgglomerativeClustering
- from sklearn.cluster import DBSCAN
- from sklearn.datasets import make_blobs
- import numpy as np
-
- # 创建虚拟数据
- centers = [[5], [0],[-5]]
- X, labels_true = make_blobs(n_samples=200, centers=centers,
- cluster_std=0.4, random_state=0)
-
- f,(ax1, ax2, ax3, ax4) = plt.subplots(1, 4, figsize=(8, 3))
- # KMeans
- km = KMeans(n_clusters=2).fit(X) # n_clusters=None, n_init=1, random_state=0
- y_km = km.labels_
- for i in range(max(y_km+1)):
- ax1.scatter(np.array([i for i in range(len(X))])[y_km==i],X[y_km==i], marker='o', s=40, label='Cluster '+str(i))
- ax1.set_title('K-means')
- ax1.set_xlabel('Feature 1')
- ax1.set_ylabel('Feature 2')
-
- # 层次聚类
- ac = AgglomerativeClustering().fit(X)
- y_ac = ac.labels_
- for i in range(max(y_ac+1)):
- ax2.scatter(np.array([i for i in range(len(X))])[y_ac==i],X[y_ac==i], marker='o', s=40, label='Cluster '+str(i))
- ax2.set_title('Agglomerative')
- ax2.set_xlabel('Feature 1')
- ax2.set_ylabel('Feature 2')
-
- # 密度聚类
- # eps 就是半径,min_samples就是 MinPts 值
- db = DBSCAN(min_samples=10).fit(X) # eps=0.2, min_samples=3, metric='euclidean'
- y_db = db.labels_
- for i in range(max(y_db+1)):
- ax3.scatter(np.array([i for i in range(len(X))])[y_db==i],X[y_db==i], marker='o', s=40, label='Cluster '+str(i))
- ax3.set_title('DBSCAN')
- ax3.set_xlabel('Feature 1')
- ax3.set_ylabel('Feature 2')
-
- # 原始数据
- ax4.scatter(np.array([i for i in range(len(X))]), X, marker='o', s=40)
- ax4.set_title('Original data')
- ax4.set_xlabel('Feature 1')
- ax4.set_ylabel('Feature 2')
-
- plt.tight_layout()
- plt.show()

因此,遇到此类数据,密度聚类效果更好。
声明:本文内容由网友自发贡献,不代表【wpsshop博客】立场,版权归原作者所有,本站不承担相应法律责任。如您发现有侵权的内容,请联系我们。转载请注明出处:https://www.wpsshop.cn/w/weixin_40725706/article/detail/774633
推荐阅读
相关标签


