热门标签
热门文章
- 1Hbuilder开发app实战-识岁01-actionsheet实例_plus.nativeui.actionsheet
- 2计算机体系结构:系统加速比计算例题(1.1 1.2 1.3)_假设在某程序的执行过程中,浮点操作时间
- 3【Python学习笔记(十)】串口被占用导致无法访问的解决办法_串口被拒绝访问python
- 4Facebook开源AI对话研究平台ParlAI ,解决人机对话最常见5类问题_人机对话国外研究
- 5在不重启MySQL的情况下用gdb工具设置变量
- 6yolov8:pt 转 onnx_yolov8 .pt转onnx
- 7新版VirtualBox设置Ubuntu与windows共享文件夹的方法_virtualbox ubuntu跟windows 系统文件共享
- 8WebView加载https页面在部分机型上(比如华为的一款机型)不能正常显示资源问题_华为手机 android webview 部分css丢失
- 9使用Docker搭建Nascab
- 10三面蚂蚁惨败,面试官要求手写算法?看完你还觉得算法不重要?_高德三面写算法吗
当前位置: article > 正文
PSP - 更新 MSA 搜索的全部 UniProt 蛋白序列库_蛋白质 msa获取
作者:AllinToyou | 2024-03-20 14:17:32
赞
踩
蛋白质 msa获取
欢迎关注我的CSDN:https://spike.blog.csdn.net/
本文地址:https://blog.csdn.net/caroline_wendy/article/details/131115471
UniProt 是一个提供蛋白质序列和功能信息的数据库,由欧洲生物信息学研究所(EMBL-EBI)、美国瑞士生物技术信息中心(SIB)和美国蛋白质信息资源(PIR)共同维护。
UniProt 包括三个主要部分:UniProt 知识库(UniProtKB)、UniProt 非冗余参考集合(UniRef)和UniProt 档案(UniParc)。
- UniProtKB 是一个高质量的注释数据库,包含两个子部分:UniProtKB/Swiss-Prot,是一个由专家手工注释的数据库,包含已知的蛋白质序列和功能信息;UniProtKB/TrEMBL,是一个由计算机自动注释的数据库,包含从基因组、转录组和蛋白组项目中收集的蛋白质序列。
- UniRef 是一个为了加快搜索速度而创建的非冗余蛋白质集合,它根据序列相似度将 UniProtKB 中的蛋白质分为三个集合:UniRef100、UniRef90 和 UniRef50。
- UniParc 是一个包含所有已知的蛋白质序列的档案,它不对序列进行任何修改或注释,只是为每个序列分配一个唯一的标识符。
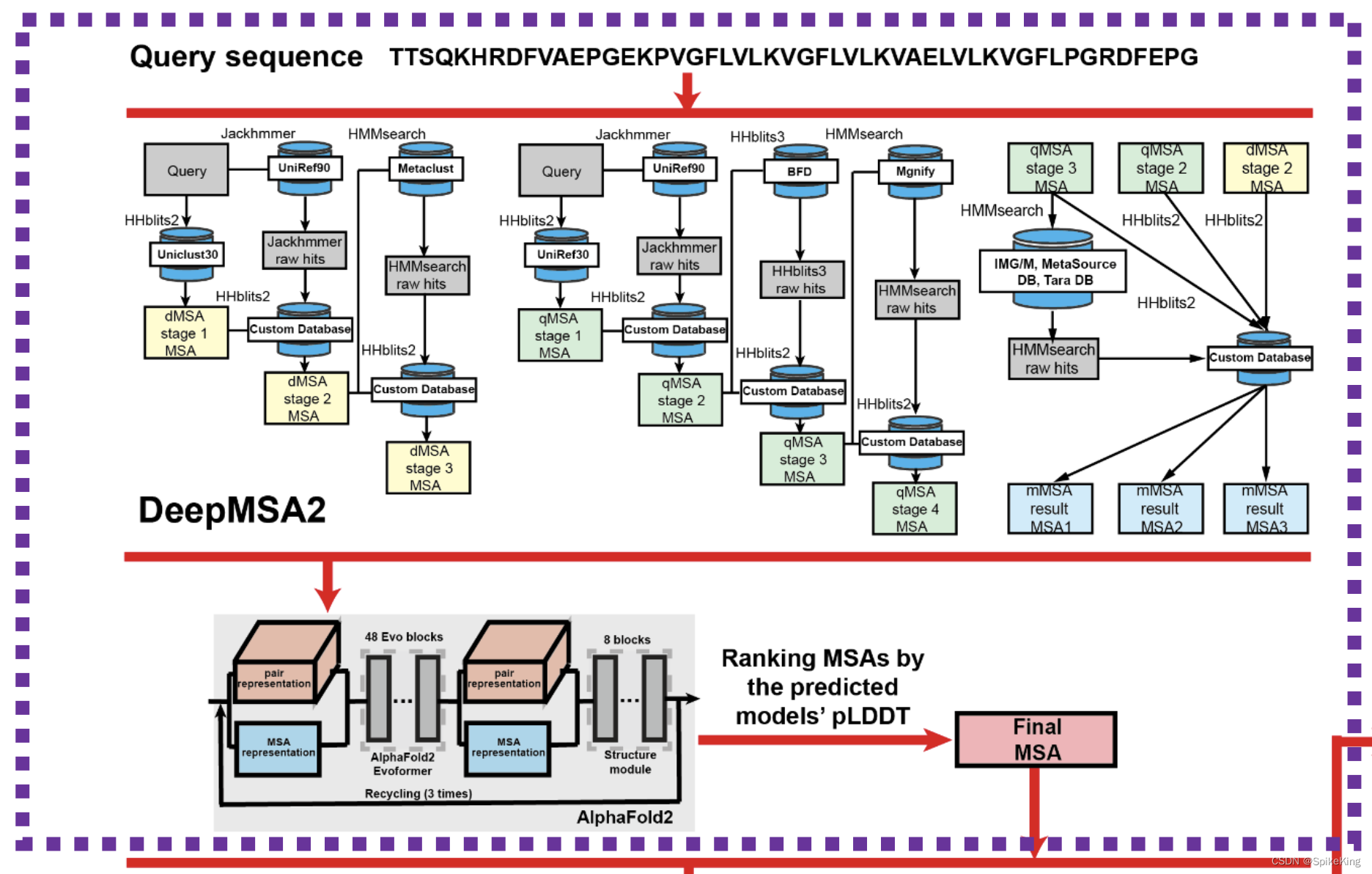
以 升级 DeepMSA2 的搜索库为例,更新 MSA 搜索的全部蛋白序列库,包括UniRef90、BFD、MGnify、UniClust30、UniRef30、MetaClust、TARA、IMG、MetaSource等,如图所示:
配置信息位于:
dit_utils/constants.py
- 1
MSA库路径的修改,如下:
# 原始路径
libdir = "[Your Folder]/ITLIB" # template library directory
# 最新路径
libdir = "[Your New Folder]/msa_databases/deepmsa2" # template library directory
- 1
- 2
- 3
- 4
- 5
工程逻辑DeepMSA2_noIMG.pl的修改,如下:
my $qhhblitsdb="$ITlibdir/uniref30/UniRef30_2022_02"; # upgrade v2: uniclust30_2017_04 -> UniRef30_2022_02
my $qjackhmmerdb="$ITlibdir/uniref90/uniref90.fasta"; # upgrade v2: file
my $qhhblits3db="$ITlibdir/bfd/bfd_metaclust_clu_complete_id30_c90_final_seq.sorted_opt"; # upgrade v2: file
my $qhmmsearchdb="$ITlibdir/mgnify/mgy_clusters_2022_05.fa"; # upgrade v2: mgy_clusters.clean.fasta -> mgy_clusters_2022_05.fa
my $dhhblitsdb="$ITlibdir/uniclust30/uniclust30_2018_08"; # upgrade v2: uniclust30_2017_04 -> uniclust30_2018_08
my $djackhmmerdb="$ITlibdir/uniref90/uniref90.fasta"; # upgrade v2: file
# upgrade v2: metaclust.fasta file; mgy_clusters.clean.fasta -> mgy_clusters_2022_05.fa
my $dhmmsearchdb="$ITlibdir/m- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
声明:本文内容由网友自发贡献,不代表【wpsshop博客】立场,版权归原作者所有,本站不承担相应法律责任。如您发现有侵权的内容,请联系我们。转载请注明出处:https://www.wpsshop.cn/w/AllinToyou/article/detail/273956?site
推荐阅读
相关标签


